R-loop是一种由RNA-DNA杂交链和一条置换出的单链DNA组成的特殊三链核酸结构,广泛存在于基因组中,并在DNA复制、转录及损伤修复等多种生物学过程中发挥关键功能【1】。细胞内R-loop的稳态需被严格调控,否则可能引发转录–复制冲突(transcription–replication conflicts,TRCs)和DNA损伤,进而威胁基因组稳定性【2】。
聚腺苷二磷酸核糖聚合酶1(poly[ADP-ribose] polymerase 1, PARP1)可利用NAD+作为底物,将ADP-核糖基团催化转移至自身或其他靶蛋白上,形成链状或分支状的多聚ADP-核糖(poly[ADP-ribose],PAR)链,从而介导下游调控因子的招募,或直接调节靶蛋白功能。通过这一机制,PARP1广泛参与DNA损伤修复与复制压力应答过程【3】。近年来,研究发现PARP1 同样参与R-loop及TRCs的调控【4】【5】。尽管已有证据表明PARP1 能够识别并结合 R-loop,但其在细胞中如何促进R-loop解离的具体机制尚不明确。由于PARP1常作为信号通路中的初始感应因子,识别协同发挥作用的下游效应因子,是揭示其调控R-loop分子机制的关键所在。
2025年6月27日,我院黄俊教授团队在Nature Chemical Biology杂志在线发表了题为The PARP1–EXD2 axis orchestrates R-loop resolution to safeguard genome stability的研究成果,系统揭示了3'–5'核酸外切酶EXD2是PARP1去除R-loop的关键下游效应因子,为阐明PARP1调控R-loop稳态及缓解TRCs的机制提供了重要支撑。

该研究通过一系列分子与生化实验揭示了EXD2与R-loop之间的紧密关联,并阐明了PARP1通过招募EXD2实现R-loop清除的分子机制。作者最终提出了一个整合性的机制模型(图1):在内源性或外源性压力诱导R-loop形成的背景下,PARP1作为初始感应因子首先识别并结合R-loop,并通过其催化合成的多聚ADP-核糖(PAR)链,招募3'–5'外切酶EXD2以及乙酰转移酶CBP至R-loop位点。随后,在CBP的催化作用下,EXD2的第416位赖氨酸残基发生乙酰化修饰,该修饰增强了EXD2对R-loop的亲和力,有效防止因PAR链快速合成与水解所引起的EXD2脱离R-loop位点。结合至 R-loop后,EXD2依赖其核糖核酸外切酶活性选择性降解R-loop中的RNA链,促进R-loop的解析,从而缓解转录–复制冲突(TRCs)并维护基因组稳定性。
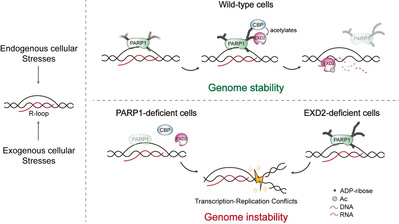
图1 PARP1-EXD2移除R-loop的分子机制模型。
当PARP1缺失或其酶活性受到抑制时,EXD2和CBP无法有效招募至R-loop位点;而在EXD2缺陷的情形下,PARP1虽可识别R-loop,但失去了其下游执行清除功能的效应因子。上述任一情况均会导致R-loop积累、TRCs加剧,最终诱发基因组不稳定性。
综上,该研究明确了EXD2在R-loop移除中的关键作用,揭示了其在维持基因组稳定性方面的全新功能。通过系统解析PARP1–EXD2信号轴,该工作阐明了细胞如何通过协同机制识别并清除R-loop,从而缓解转录–复制冲突(TRCs)并维持基因组完整性。作者构建了一个完整的机制模型,呈现了R-loop从被识别到被精准移除的动态过程,不仅为理解R-loop相关疾病的发生机制提供了新的视角,也为靶向R-loop的疾病干预策略提供了潜在的分子靶点。此外,该研究还为PARP抑制剂的优化和新一代药物的开发提供了理论依据与新思路。
我院博士研究生李兆爽、刘雨以及华中科技大学的刘源辉博士为该文章的共同第一作者;浙江大学黄俊教授、刘婷教授、方东教授以及周建维教授为本文的共同通讯作者。香港大学的Michael Shing Yan Huen教授、我院的陆华松教授对本研究提供了帮助。
原文链接:https://www.nature.com/articles/s41589-025-01952-x
参考文献
1. Petermann E, Lan L, Zou L. Sources, resolution and physiological relevance of R-loops and RNA-DNA hybrids. Nat Rev Mol Cell Biol. 2022 Aug;23(8):521-540. 2. Brickner JR, Garzon JL, Cimprich KA. Walking a tightrope: The complex balancing act of R-loops in genome stability. Mol Cell. 2022 Jun 16;82(12):2267-2297.3. Ray Chaudhuri A, Nussenzweig A. The multifaceted roles of PARP1 in DNA repair and chromatin remodelling. Nat Rev Mol Cell Biol. 2017 Oct;18(10):610-621.4. Laspata N, Kaur P, Mersaoui SY, Muoio D, Liu ZS, Bannister MH, Nguyen HD, Curry C, Pascal JM, Poirier GG, Wang H, Masson JY, Fouquerel E. PARP1 associates with R-loops to promote their resolution and genome stability. Nucleic Acids Res. 2023 Mar 21;51(5):2215-2237.5. Petropoulos M, Karamichali A, Rossetti GG, Freudenmann A, Iacovino LG, Dionellis VS, Sotiriou SK, Halazonetis TD. Transcription-replication conflicts underlie sensitivity to PARP inhibitors. Nature. 2024 Apr;628(8007):433-441.